Serotipado de aislados de campo de Actinobacillus pleuropneumoniae mediante PCR
07-jun-2019 (hace 5 años 10 meses 11 días)
Determinar el serotipo de Actinobacillus pleuropneumoniae (App) es importante para su control, ya que los distintos serotipos tienen diferente potencial de virulencia (dependiendo de la zona geográfica), y esta información se puede utilizar para elegir la vacuna más apropiada (Gottschalk, 2015). En base a su cápsula, hay 18 serotipos conocidos de App (Bossé et al., 2018a), siendo 1, 5, 9 y 11 los más virulentos. En base a los requerimientos de NAD (nicotinamida adenina dinucleótida), se diferencian los aislados del biotipo 1 de los del biotipo 2, o NAD-independentes.
Clásicamente, para determinar el serotipo se han utilizado pruebas serológicas que incluyen la aglutinación, la coaglutinación, la inmunodifusión, la hemaglutinación indirecta y la precipitación en anillo. Sin embargo, se requieren antisueros específicos con títulos altos, y las reacciones cruzadas entre serotipos (p.e. 3/6/8 y 1/9/11) son un problema. Encontramos que la mayoría de los aislados del Reino Unido e Irlanda asignados serológicamente como serotipo 3 pertenecían al serotipo 8 (O'Neill et al., 2010). Debido a estos problemas, hemos desarrollado pruebas moleculares usando la reacción en cadena de la polimerasa (PCR) para amplificar secuencias de ADN específicas de cada serotipo que se encuentran en los genes de biosíntesis de la cápsula (Bossé et al., 2014). El uso de estas secuencias específicas de cada serotipo permitió desarrollar un test específico para el serotipo 16 (Bossé et al., 2017), y condujo al reciente descubrimiento de los serotipos 17 y 18 (Bossé et al., 2018a). Con el descubrimiento de los serotipos 16-18 decidimos producir un test de PCR que pudiera asignar todos los serotipos conocidos de App. Sin embargo, los serotipos 9 y 11 no pueden diferenciarse mediante PCR, ya que sus loci capsulares son prácticamente idénticos (Bossé et al., 2018b), y tampoco pueden determinarse mediante tests serológicos (Gottschalk, 2015). El test de PCR tenía que confirmar el aislado como App y determinar el serotipo específico. Para confirmar App, utilizamos primers que amplificaban una región de 418 bp del gen apxIV, específico de App (Schaller et al., 1999). Sin embargo, debido al gran número de serotipos (n=18), tuvimos que formular dos PCR multiplex (mPCR), capaz cada uno de los cuales de detectar muchos serotipos. El mPCR1 detecta los serotipos 1-12 y 15 (figura 1A) y el mPCR2 los serotipos 13-14 y 16-18 (figura 1B). Los aislados que amplifican sólo una banda apxIV en el mPCR1 son analizados después con el mPCR2.
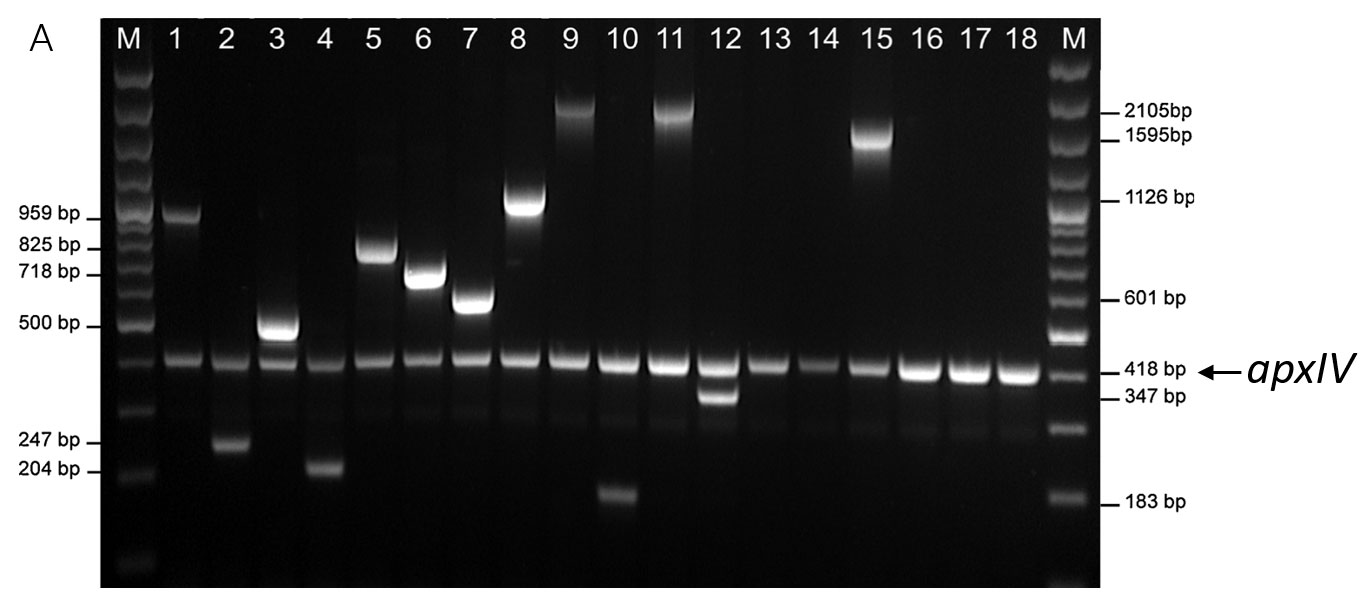
![]()
mPCR2 también confirma el biotipo mediante cebadores diseñados para amplificar un fragmento de 1339 bp del gen nadV (que confiere la independencia al NAD). El amplicón nadV de 1339 bp sólo se detecta en las cepas de referencia del biotipo 2 (serotipos 13-14). Cabe señalar que otros serotipos (p.e. 2, 4, 7 y 17) han sido descritos como pertenecientes al biotipo 2, y algunos serotipos de aislados norteamericanos del serotipo 13 como pertenecientes al biotipo 1 (Gottschalk, 2015).
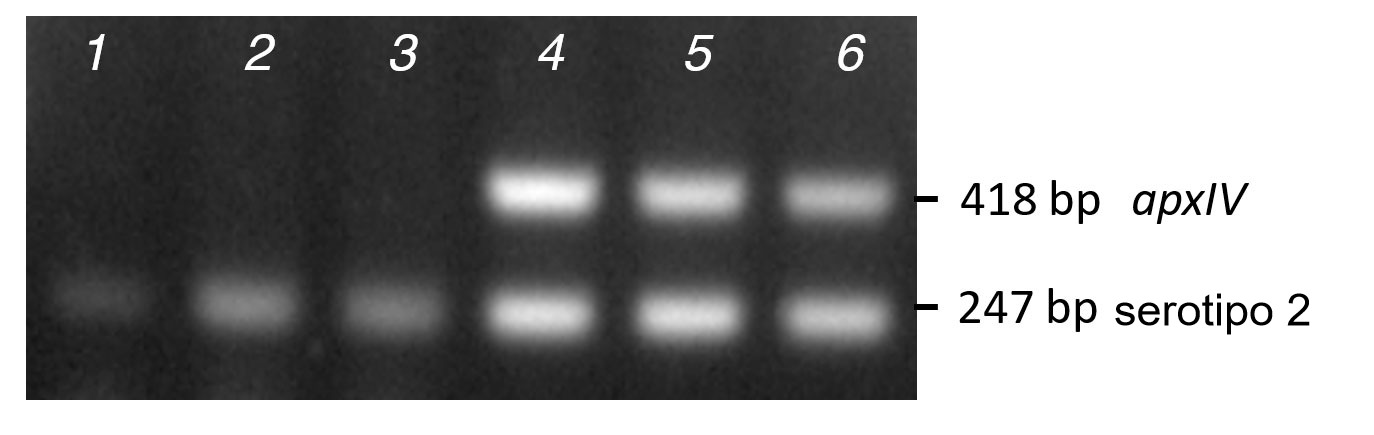
La plantilla de ADN para las PCRs puede ser ADN purificado (ya sea de bacterias cultivadas o de muestras de tejidos) obtenido usando kits comerciales, lisados de células bacterianas enteras hervidas o colonias de un cultivo en placa. El ADN purificado proporciona mejores resultados, mientras que las PCRs de colonias pueden dar resultados parciales/falsos negativos si las colonias son muy pegajosas y difíciles de lisar, como muestran los tres aislados clínicos de serotipo 2 que sólo amplificaron la banda específica de serotipo de PCR de colonia, pero que amplificaron tanto esta como la banda específica de apxIV al usar ADN purificado (figura 2). En raras ocasiones, los aislados no lograrán amplificar apxIV, incluso utilizando ADN purificado (Bossé et al., 2014). Cuando esto sucede, pueden utilizarse primers alternativos de apxIV (oAPXIV-TSP1/2) para confirmar App (Tegetmeyer et al., 2008). mPCR1 y mPCR2 pueden usarse para identificar nuevos serotipos, como hicimos con los 17 y 18 (Bossé et al., 2018a). Los aislados de App que producen una banda de apxIV pero no amplicones específicos de serotipo pueden, potencialmente, ser un serotipo nuevo, aunque la ausencia de una banda específica de serotipo puede ser debida a una falta de concordancia del primer en aislados divergentes o deberse a la presencia de un elemento de inserción que altera el locus de la cápsula. La secuenciación del genoma completo del aislado confirmará cuál de estas opciones es la correcta.
En resumen, la monitorización de serotipos de APP en una granja o país es importante para controlar la enfermedad. Nuestros PCRs de serotipado (mPCR1 y mPCR2) son herramientas útiles para identificar serotipos virulentos, las vacunas correctas a utilizar (comerciales o autógenas), y para evitar que cerdos con cepas de App potencialmente virulentas sean introducidos en granjas no expuestas. Además, nuestro método tiene el potencial de identificar nuevos serotipos de App y mejorar los diagnósticos.
Agradecimientos
La investigación sobre APP en el laboratorio de los autores cuenta con el apoyo del Consejo de Investigación de Biotecnología y Ciencias Biológicas del Reino Unido.
