Limitaciones de la PCR en el diagnóstico del PRRS
29-nov-2018 (hace 6 años 4 meses 2 días)
Los dos métodos de diagnóstico más utilizados para detectar el PRRS son ELISA y PCR. En este artículo nos centraremos en la PCR, el método con el que se puede detectar el virus en prácticamente cualquier material. Se basa en la polimerización de ADN cíclico de un ADN o, como en el caso del virus del PRRS, fragmentos de ADNc flanqueados por secuencias cortas a las que se unen cebadores de ADN específicos en cada ciclo de PCR. La sensibilidad y la especificidad de un ensayo de diagnóstico en el que se emplea la PCR depende de la idoneidad de estos cebadores para su objetivo y los ciclos térmicos empleados para la amplificación cíclica. Lo ideal sería que fueran complementarios solo para el genoma de una especie de virus concreta y que no fueran capaces de unirse a ningún otro microbio o secuencia de ADN hospedador siendo, aún así, capaces de detectar genomas de todas las cepas o variantes de esa especie de virus. Por lo tanto, su objetivo son los fragmentos más conservados del genoma.
El virus del PRRS es uno de los patógenos para los cuales es más difícil desarrollar tests PCR altamente sensibles y específicos. Los virus del PRRS conocidos actualmente pertenecen a dos especies, PRRSv-1 y PRRSv-2. Ambas especies de virus del PRRS se propagan a nivel mundial, pero el PRRSv-1 predomina en Europa, mientras que el PRRSv-2 predomina en América y Asia. Se puede suponer que la diversidad genética de la especie de virus predominante es mayor que la del que tiene una prevalencia menor. En efecto, los ensayos de PCR que se utilizan, por ejemplo, en Europa o América, pueden validarse mejor y ser más adecuados para detectar PRRSv-1 o PRRSv-2, respectivamente. Como consecuencia, algunas cepas de la especie «secundaria» en un área determinada, o un continente, podrían no detectarse, con lo que se subestimaría su papel en la patogenia del PRRS. Esto es particularmente importante en aquellas granjas donde conviven PRRSv-1 y PRRSv-2. La gran diversidad genética de los virus del PRRS requiere una actualización constante de las secuencias de los cebadores y las sondas de PCR para seguir la evolución continua del virus y los descubrimientos de variantes genéticas completamente nuevas. Esto es especialmente cierto para el PRRSv-1, cuya diversidad genética extrema, unida a la baja disponibilidad de virus de referencia de Rusia, Bielorrusia y Ucrania, amenaza con afectar la sensibilidad de los kits de PCR comerciales para los subtipos genéticos del PRRSv-1 en Europa del Este.
Actualmente, la variante de PCR en tiempo real es la utilizada principalmente en el diagnóstico de rutina. Existen varios métodos de PCR en tiempo real que difieren en sus principios, pero el más utilizado es el método con las llamadas sondas TaqMan. Una sonda TaqMan es un fragmento de ADN corto (oligonucleótido) marcado con fluoróforos, que se une a la diana de ADN entre los cebadores flanqueantes. Si se produce la unión, la sonda se lisa durante la polimerización del ADN y el instrumento de PCR registra la fluorescencia. La PCR en tiempo real es particularmente adecuada para la multiplexación de los objetivos del diagnóstico. Esto significa que en una única reacción de PCR (o en una única prueba) se pueden detectar 2-3 patógenos diferentes a través de la emisión de fluorescencia de 2-3 tintes diferentes. Por lo tanto, ambas especies de virus del PRRS pueden detectarse al mismo tiempo fácilmente y diferenciarse en muestras de un solo animal, corral o granja.
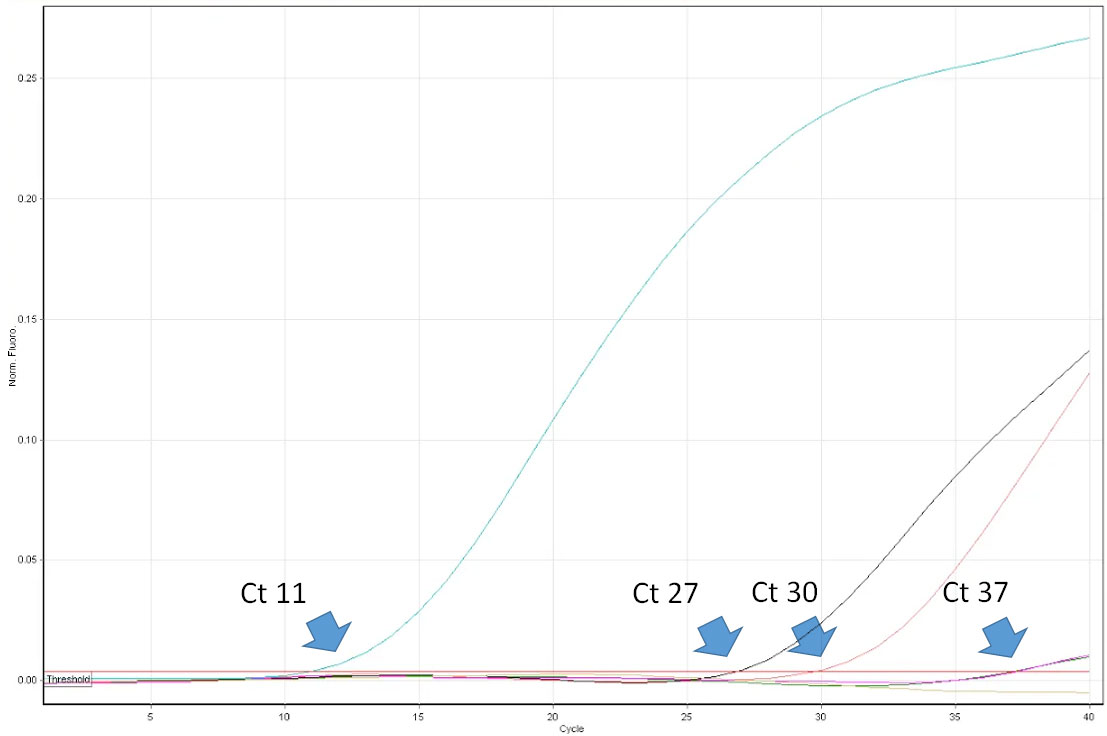
La interpretación de los resultados de la PCR es engañosamente sencilla. El veterinario debe conocer las limitaciones del método y ser capaz de interpretar críticamente los resultados. Por ejemplo, un resultado positivo en la PCR demuestra la presencia de un ácido nucleico diana (p. ej., del virus del PRRS) en la muestra. Sin embargo, no prueba que la muestra contenga virus infecciosos. La PCR en tiempo real permite la cuantificación del virus en la muestra. Sus resultados se expresan como «ciclo umbral» (Ct), que es el primer número de ciclo en el que el instrumento de PCR detectó la emisión de fluorescencia desde el tubo de reacción. Cuanto menor sea el número de ciclo, mayor será el número de copia del ADN objetivo (leído como número de copias de virus). Por ejemplo, una muestra con Ct 20 puede considerarse altamente positiva, y una muestra con Ct 35 un positivo bajo. Sin embargo, en algunos casos la fluorescencia se detecta solo en ciclos muy tardíos de PCR en tiempo real, p. ej., >38. ¿Cómo se pueden interpretar valores tan altos? Lamentablemente, ese resultado no siempre indica la presencia de un número muy bajo de copias de virus en la muestra. Los denominados «virus de crecimiento tardío» pueden ser el resultado de la ruptura espontánea de una sonda después de muchos ciclos térmicos de la PCR, que simulan la nucleólisis enzimática causada por la ADN polimerasa. Por lo tanto, puede suceder también en ausencia del ácido nucleico del virus en la muestra. La importancia de la correcta interpretación de la presencia de «virus de crecimiento tardío» es máxima al monitorizar las poblaciones sin virus del PRRS. Debe realizarse un diagnóstico de seguimiento, incluyendo la técnica ELISA. Desgraciadamente, algunos laboratorios de diagnóstico en Europa no proporcionan a los clientes valores de Ct precisos de PCR en tiempo real. Al contrario, los resultados se expresan solo como negativos o positivos. La utilidad de dichos informes para los profesionales es limitada y, en algunos casos, puede dar lugar a decisiones erróneas con respecto a los protocolos de control del virus del PRRS.
Consideraciones prácticas
- Ninguno de los métodos actuales de PCR puede detectar todos los virus PRRS.
- El resultado negativo de una PCR de unas cuantas muestras aleatorias de una granja no demuestra la negatividad a PRRS.
- El resultado positivo de una PCR demuestra la presencia de ácido nucleico de PRRSv en una muestra, pero no necesariamente su infectividad.
