Virus de la influenza porcina tipo A : situación en Europa
16-dic-2016 (hace 8 años 3 meses 16 días)
Las infecciones por el virus de la influenza A (IAV) presentan una morbilidad significativa en las poblaciones porcinas de todo el mundo. La infección se relaciona básicamente con síntomas respiratorios agudos que frecuentemente van acompañados de co-infecciones bacterianas que pueden desencadenar un aumento de la mortalidad en cerdos jóvenes, una disminución del crecimiento en cebo o abortos en gestantes. Por todo ello, las pérdidas económicas pueden ser considerables (Rajao et al., 2014; Vincent et al., 2014; Vincent et al., 2014).
Los virus influenza A porcinos pertenecen a la familia Orthomyxoviridae; son virus RNA de cadena simple cuyo genoma consta de 8 segmentos. Los subtipos se clasifican según las características genéticas y antigénicas de las glicoproteínas de superficie hemaglutinina (HA) y neuraminidasa (NA) (Cheung et al., 2007).
Los grupos de glicanos con ácidos siálicos terminales actúan como receptores para la adhesión del IAV a las células superficiales del tracto respiratorio. La expresión de receptores con ácidos siálicos en α-2-6 y α-2-3 en el tracto respiratorio superior de los cerdos hace que sean susceptibles a virus influenza A tanto de origen aviar como humano (Byrd-Leotis et al., 2014; Trebbien et al., 2011; Walther et al., 2013). Por tanto, las co-infecciones de células porcinas con IAV aviares y/o humanos abren la posibilidad de que se produzcan reordenaciones genéticas (intercambio de fragmentos de genoma) entre estos virus (Hass et al., 2011; Ma et al., 2009; Van Poucke et al., 2010; Zell et al., 2013). Tales reordenaciones pueden suponer cambios fenotípicos inesperados o difíciles de predecir que afecten al tropismo de huéspedes y tejidos, a la virulencia y estructura antigénica, en comparación con los virus parentales.
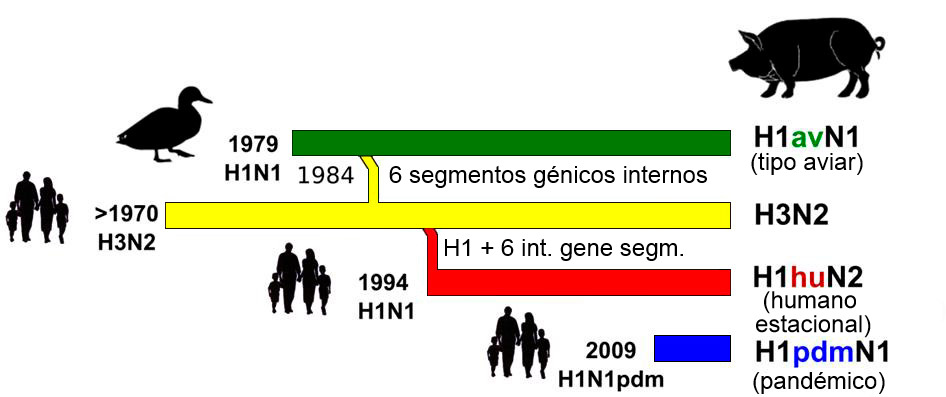
La figura 1 muestra el origen y la historia de la reordenación de los IAV porcinos que circulan actualmente en las poblaciones porcinas europeas. Hasta 2009, en Europa circulaban tres subtipos de IAV, H1N1, H1N2 y H3N2. El linaje más antiguo, el H1N1, se introdujo en la población porcina a finales de los 70 a partir de un origen aviar y reemplazó al linaje H1N1 tipo clásico derivado del humano de 1918 que estaba circulando en los años previos. Al ser un virus influenza de origen aviar, al linaje H1N1 posterior a los 70 se le denomina H1N1 "tipo aviar" (o "av", del inglés "avian-like") (Brown, 2013; Simon et al., 2014). En 1984, se produce una reordenación entre un virus estacional humano H3N2 y el subtipo H1avN1av cuyo virus resultante contiene todos los 6 fragmentos internos de origen aviar, y los HA (H3) y NA (N2) del virus humano (Kong et al., 2015). Este nuevo virus se establece como endémico en las poblaciones de cerdos de varios países europeos pero aparentemente no en todos. En 1994, una nueva reordenación afectó al virus H3N2 porcino, por la cual este virus intercambió su fragmento genómico HA con el de otro virus estacional humano H1N1 para generar el linaje H1N2. Este subtipo H1 se denomina H1N2 "tipo humano" (o "hu") (Brown, 1998). Estos linajes circularon con una relativa estabilidad en los países europeos con algunas restricciones geográficas respecto a su prevalencia (Brown, 2013; Kuntz-Simon et al., 2009). Los virus del linaje H1N1av parecían ser los responsables de la mayoría de infecciones en cerdos en Europa (Brown, 2000).
En 2009, un cuarto actor entra en juego: el virus influenza A H1N1 pandémico de 2009 (H1N1pdm). Este virus es el resultante de múltiples reordenaciones y está constituido por fragmentos originados a partir de virus americanos tipo aviar (PB2, PA), humano (PB1) y porcino clásico (HA, NP, NS), y virus europeos tipo porcino (NA, NP) (Neumann et al., 2009). En referencia al origen porcino, por lo menos parcialmente, de esta última pandemia de gripe, la infección en humanos se ha llamado "gripe porcina". Los cerdos parecían ser muy susceptibles a este tipo de IAV y la transmisión zoonótica inversa de humanos infectados a cerdos se registró en varios sitios en todo el mundo. Desde entonces se producen infecciones por H1N1pdm en poblaciones porcinas en ciclos independientes de las infecciones en humanos (Brookes et al., 2010).
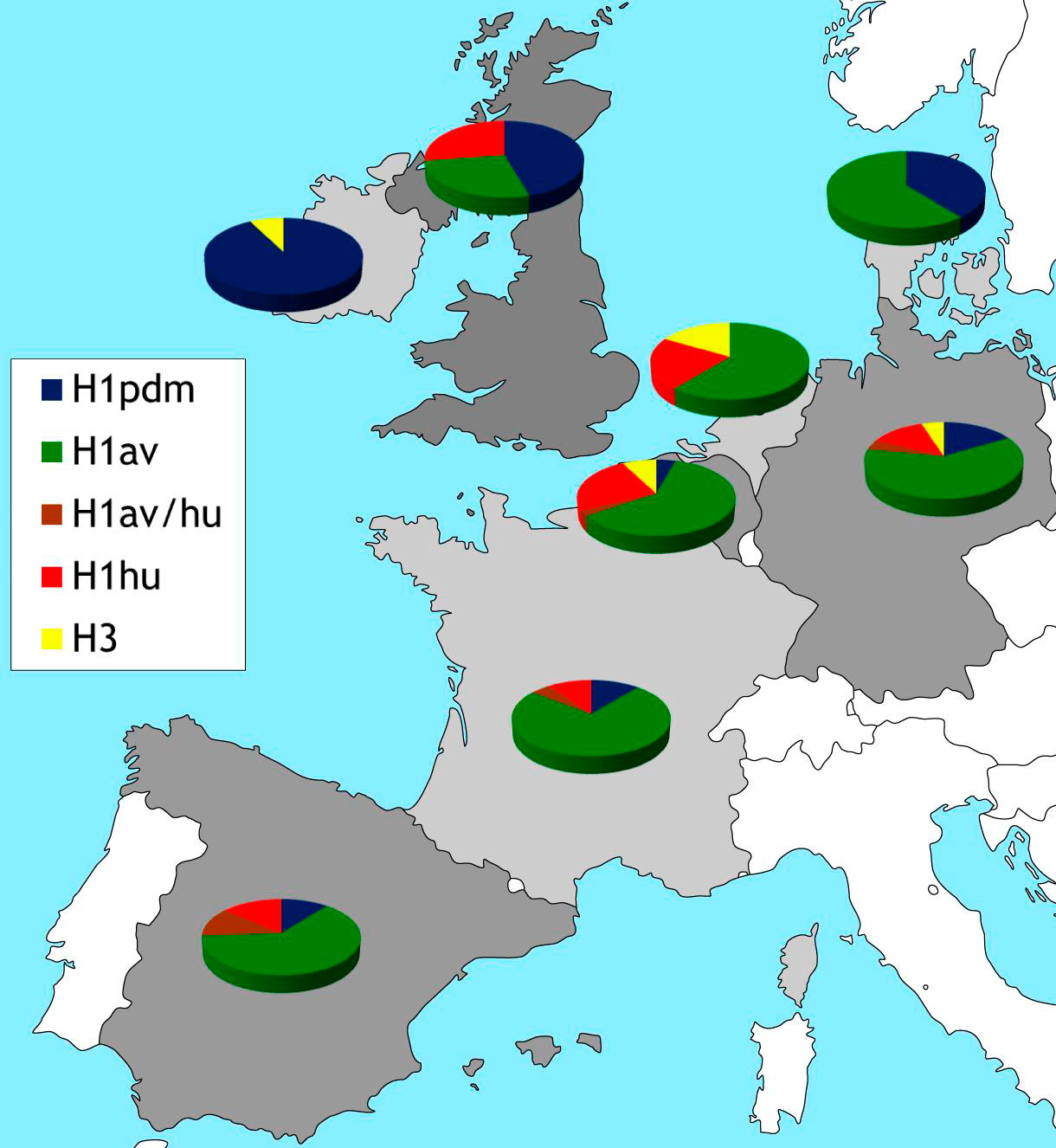
A raíz de lo que se denominó como “gripe porcina” se llevó a cabo un programa de vigilancia para la influenza porcina en Europa desde 2013 a 2105, a través del proyecto ESNIP3 (Watson et al., 2015). Los resultados mostraron que el H1N1dpm está presente en varios países europeos, y en particular con una elevada prevalencia en Gran Bretaña e Irlanda. La presencia de H1N1dpm de origen humano en las poblaciones porcinas europeas obviamente alteró el fino equilibrio entre los tres auténticos linajes de virus influenza porcinos europeos y conllevó el aumento de eventos de reordenación. Entre los virus emergentes derivados de las reordenaciones de los fragmentos H y/o N, en Alemania surgió el linaje H1pdmN2, el cual ya lleva varios años circulando con baja prevalencia (Starick et al., 2012; Watson et al., 2015). La proteína HA de este virus emergente ha sufrido cambios significativos y se puede diferenciar antigénicamente del H1pdm de origen humano (Harder et al., 2013). Se han descrito otras reordenaciones (H1huN1av, H1dpmN1av, H3N1pdm) que no han sido capaces de mantenerse en circulación (Watson et al., 2015). Además, también se han descrito reordenaciones que han intercambiado todos o algunos de seis los fragmentos internos del genoma con virus H1N1pdm (Kong et al., 2015; Watson et al., 2015). En USA, los virus H3N2 o H1N1 portadores del fragmento M del H1N1pdm se asociaron con una mayor transmisión del porcino a humanos en ferias agrícolas (Schicker et al. 2016). Estas “variantes” de virus también se han descrito esporádicamente en Europa pero no se han asociado a infecciones en humanos. La prevalencia del H1N1dpm en las poblaciones porcinas europeas varía considerablemente (figura 2). A diferencia de lo que sucede en las islas Británicas, el subtipo porcino H1N1av todavía cuenta con la mayor prevalencia en la Europa continental mientras que los subtipos H3N2 y H1huN2 presentan prevalencias menores (figura 2). El subtipo H3N2 no se ha detectado en Dinamarca, Polonia o el Reino Unido, y sólo unos pocos brotes esporádicos se han reportado en Francia (Watson et al. 2015).
Varios países de Europa central y del este empezaron estudios de monitorización pasiva cuando terminó el proyecto ESNIP3 en 2015 que se han realizado en nuestro laboratorio. Los datos preliminares del primer año de monitorización revelan un aumento de la frecuencia del H1N1pdm y sus reordenaciones. Además, se evidencia una mayor frecuencia de co-infecciones y más actividad de reordenación entre los tres linajes H1 porcinos europeos que se encuentran circulando. No está muy claro en qué medida estos hallazgos están sesgados por la implementación de herramientas de diagnóstico preciso desarrolladas por nuestro laboratorio: la detección de IAV porcino y la caracterización de linajes y subtipos HA (H1av, H1hu, H1pdm, H3) y NA (N1av, N1pdm, N2) se consigue a través de un set de tres RT-qPCR en tiempo real. La mayor sensibilidad de estas pruebas permite un análisis directo sobre muestras de campo (en particular hisopos nasales) de forma independiente al aislamiento vírico en los cultivos celulares (Henritzi et al. 2016). Estos estudios continuarán al menos hasta finales de 2017.
La continua monitorización de las poblaciones porcinas en Europa es esencial para seguir la evolución del IAV porcino. Dicho virus puede jugar un papel importante en la formación de virus pandémicos en humanos tal como se ha demostrado por la aparición del virus H1N1 2009. El mejor conocimiento de los cambios genotípicos constantes y de la aparición de nuevos fenotipos de IAV con mayor propensión zoonótica es la clave de la política "OneHealth".
Agradecimientos
Los estudios recientes sobre IAV porcino en el laboratorio de los autores han sido financiados por IDT, Dessau, Alemania. Los autores agradecen el excelente soporte técnico brindado por Aline Maksimov and Carl Sell.
