Utilidad de la PCR en Tiempo Real en el diagnóstico de las colibacilosis porcinas
25-may-2015 (hace 9 años 10 meses 30 días)
Escherichia coli forma parte de la flora intestinal del cerdo y bajo determinadas circunstancias produce cuadros de colibacilosis, una enfermedad que puede manifestarse bajo diferentes procesos tales como diarrea neonatal, diarrea postdestete, enfermedad de los edemas, septicemia, etc…
Un adecuado diagnóstico comprende una exhaustiva anamnesis, una correcta selección de muestras y un completo diagnóstico diferencial apoyado por diversas técnicas. La mera detección de cepas patógenas no justifica siempre la enfermedad dado que es habitual encontrar portadores asintomáticos. La identificación de cepas relevantes por su virulencia y su diferenciación del resto de la flora habitual del intestino es un reto diagnóstico de plena actualidad y la PCR en tiempo real (qPCR) aplicada a la detección de los factores de virulencia (FV) de E. coli se presenta como una potente herramienta capaz de generar valiosa información.
Partiendo de la extracción de los ácidos nucleicos de diferentes matrices biológicas (cultivo, heces, intestino o hisopado rectal), los ensayos qPCR reconocen regiones específicas del genoma de E. coli que codifican distintos FV. En este trabajo, cada ensayo fue diseñado para la detección de un FV distinto (ver tabla 1). Una qPCR adicional (ECCO) fue utilizada para confirmar la presencia de E. coli en las muestras y asegurar que todas las etapas de la técnica se han realizado correctamente. La qPCR tiene carácter cualitativo pero también cuantitativo (Imagen 1), ventaja notable frente a su predecesora, la PCR convencional.
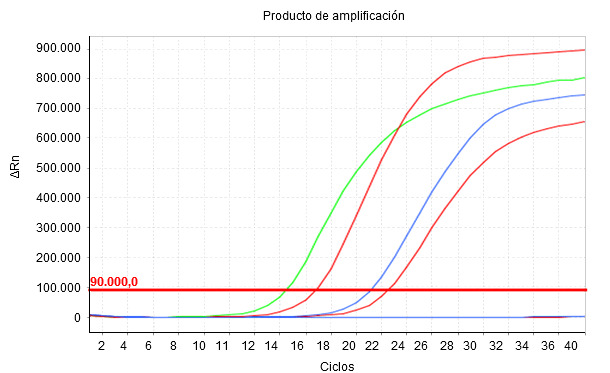
Imagen 1. El producto de amplificación se muestra en tiempo real a medida que transcurren los ciclos térmicos (cicles) de tal manera que el nivel de fluorescencia registrada (ΔRn) aumenta exponencialmente. El Cq es el número de ciclos en el que la fluorescencia empieza a ser detectada. Cuanto menor es el valor Cq, mayor es la concentración inicial del parámetro estudiado en la muestra.
La evaluación de cepas virulentas en los procesos de colibacilosis se ha realizado tradicionalmente mediante un cultivo microbiológico inicial, aislamiento de colonias de E. coli y la posterior caracterización de sus FV. Esta metodología presenta el inconveniente de un análisis limitado al número de aislamientos seleccionados confiando la exactitud del diagnóstico a la representatividad de éstos dentro del total de la población de E. coli en la muestra estudiada. De esta manera, se corre el riesgo de pasar inadvertidas poblaciones no mayoritarias de E. coli que sin embargo sí tengan relevancia clínica.
La qPCR ofrece la posibilidad de realizar el análisis de los FV directamente sobre la muestra clínica. Esta metodología evita etapas intermedias de cultivo y las respectivas determinaciones sobre cada uno de los distintos aislados seleccionados, lo que conlleva un ahorro notable de tiempo y costes. Dado el carácter cuantitativo de la técnica se puede establecer una relación entre el número de copias detectado para un determinado gen de virulencia y la población total de E. coli presente en la muestra. De este modo podríamos interpretar la posibilidad de conseguir aislar una cepa con una combinación particular de FV.
Esta metodología presenta ciertas limitaciones debido al indeterminado número de copias de los distintos genes que codifican cada FV dentro de diferentes poblaciones bacterianas. No obstante, cumple el objetivo propuesto: valorar la posibilidad de encontrar en la muestra una población de E. coli con una determinada combinación de FV.
A continuación exponemos un ejemplo práctico. Ante un caso de sospecha clínica de enfermedad de los edemas en animales de cebo, se remitieron al laboratorio cuatro intestinos delgados (ID) pertenecientes todos ellos a animales afectados para valorar la presencia de E. coli verotoxigénico. El pool formado por los 4 intestinos se analizó directamente mediante qPCR, resultando unos valores Cq para F18, Stx2e y ECCO casi coincidentes (Tabla 1). Estos resultados se pueden interpretar como la presencia de una población ampliamente mayoritaria de E. coli que posee los genes codificantes para F18 y Stx2e, es decir, una cepa verotoxigénica. Además se obtuvo un valor débilmente positivo para F4 (Cq=36,9) lo que indicaría una presencia muy minoritaria de poblaciones con dicho FV.
Simultáneamente se realizó un cultivo microbiológico a las muestras en el que 12 colonias de E. coli fueron seleccionadas. Su comparación fenotípica reveló la presencia de una única cepa (Imagen 2). La caracterización de la misma mediante determinación de sus FV por qPCR confirmó que se trataba de una potencial cepa verotoxigénica ya que fueron detectados simultáneamente los genes codificantes para F18 y Stx2e.
Tabla 1. Resultados qPCR.
| FV | Pool 4 intestinos delgados | Cepa E. coli aislada |
| F4 | POS (Cq = 36,9) | neg |
| F5 | neg | neg |
| F6 | neg | neg |
| F18 | POS (Cq = 22,28) | POS (Cq = 16,58) |
| F41 | neg | neg |
| LT | neg | neg |
| STa | neg | neg |
| STb | neg | neg |
| Stx2e | POS (Cq = 22,39) | POS (Cq = 17,29) |
| eae | neg | neg |
| ECCO | POS (Cq = 21,1) | POS (Cq = 16,97) |
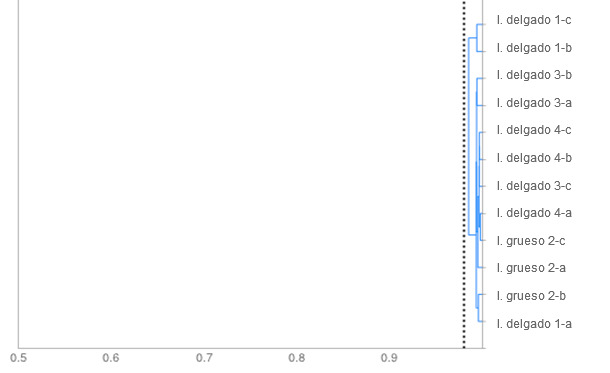
Imagen 2. Dendograma. Se muestra el grado de similitud entre los aislamientos de E. coli en función del patrón de fermentación de azúcares. Todos los aislamientos guardan una similitud superior al punto de corte establecido en 0,98 (línea de puntos); lo que nos hace concluir que se trata de una única cepa.
En conclusión, la qPCR aplicada directamente a muestras clínicas puede suponer un ahorro sustancial de recursos y tiempo ofreciendo una información capaz de llevarnos a una interpretación que aporte soluciones al delicado diagnóstico de las colibacilosis porcinas.
