Resistencia Genética al PRRS
20-sep-2010 (hace 14 años 6 meses 12 días)
Aunque el PRRS es una enfermedad relativamente nueva, que apareció por primera vez en Canadá en 1979, ahora es endémica en casi todos los países del mundo y causa pérdidas económicas devastadoras debido a la reducción de la producción de lechones, problemas de reproducción y trastornos respiratorios. Varios cálculos sugieren que entre un cuarto y un tercio del coste atribuido a enfermedades infecciosas se debe al PRRS. A menudo la enfermedad es persistente, de manera que que el animal se recupera pero continúa infectando a los cerdos no expuestos. Esto sugiere que puede haber una producción tardía de anticuerpos neutralizantes, una reducción de la respuesta inmunitaria tanto innata como adquirida, y una mutación alta del virus PRRS durante la replicación, aumentando su diversidad genética.
Una colaboración internacional coordinada está investigando varios aspectos sobre el PRRS, pero el “santo grial” de producir cerdos genéticamente resistentes está resultando bastante elusivo. Sin embargo, las herramientas y tecnologías desarrolladas en los proyectos ‘genoma’ están ofreciendo nuevas oportunidades para desentrañar los componentes genéticos de la respuesta a la infección de PRRS.
Existen amplias evidencias de una variación significativa de la respuesta y de la tolerancia entre razas. Por ejemplo, estudios alemanes han mostrado que, tras una exposición a PRRS, una linea de cerdo miniatura mostró una duración reducida de la viremia y la persistencia, y también una carga viral considerablemente más baja que una línea comercial de Pietrain. En los EEUU, se demostraron diferencias muy significativas en la respuesta de cerdos Duroc-Hampshire respecto a Large White-Landrace.
Un estudio colaborativo reciente del Instituto Roslin de Edimburgo fue el primer estudio SNP de todo el genoma que se preguntó “¿puede detectarse una parte de la variación genética por marcadores genéticos?” Los investigadores analizaron datos acumulados de más de 8000 camadas en una sola explotación a través de un periodo de 10 años durante el cual fueron reportados dos brotes de PRRS. Mediante un chip SNP de 7K se identificaron varios SNP significantes actuando independientemente, lo cual sugiere que hay muchas regiones genéticas que regulan la tolerancia a PRRS. Los genotipos incluyen Landrace, Large White, Pietrain, Meishan, Duroc y varios cruces. Curiosamente, el impacto del virus PRRS fue mayor en la raza Meishan que en sus equivalentes europeos. En otras explotaciones se está llevando un estudio utilizando un chip SNP más grande y actual para ver si los SNP identificados siguen siendo significantes entre explotaciones y también para verificar si hay algunos efectos adversos correlacionados sobre las características productivas.
Mientras tanto, en Italia, se ha recopilado una base de datos que combina muestras de ADN con datos genealógicos y de productividad con datos sanitarios y de producción. Para ello se han utilizado más de 5000 animales de cuatros genotipos (Duroc, Landrace, Large White y Pietrain) de 18 explotaciones genéticamente relacionadas. Se identificaron varios SNP muy significativos, y el trabajo continúa investigando varios marcadores y si estos son consistentes a lo largo de las pruebas.
Representación de escalamiento multidimensional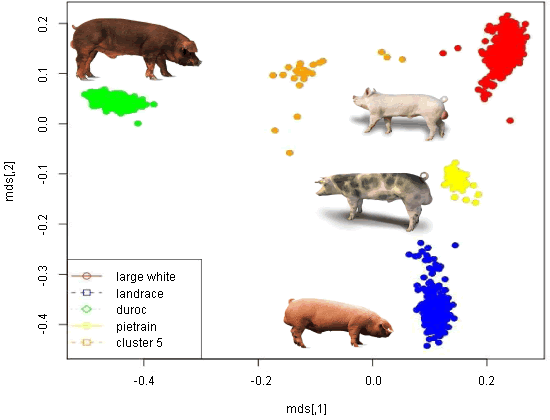 Relación genómica – Escalamiento multidimensional (MDS): distribución de clusters SNP por razas. |
Los resultados de las investigaciones hasta el momento sugieren que hay suficientes variaciones genéticas en la respuesta a la infección para esperar mejoras en la resistencia o en la tolerancia por selección, por lo que hay razones para creer que seria posible producir animales comerciales con mayor resistencia o tolerancia a la enfermedad. Sin embargo, la complejidad de esta enfermedad y el alto número de marcadores potencialmente involucrados sugiere que la rentabilidad de la producción de estos animales en grandes cantidades no podrá ser posible hasta dentro de unos años.
Definiciones:
Un chip SNP es un pequeño trozo de silicona que contiene muchas ‘sondas’ de ADN. Estas se adhieren selectivamente a las moléculas de ADN cuyas secuencias de aminoácidos son completamente complementarias – a cada T le corresponde una A y a cada G una C. Pueden ser utilizados para identificar la presencia de secuencias especificas de ADN y a buscar secuencias de ADN que difieren en ‘polimorfismos nucleotídicos simples’.
SNP (pronunciada SNIP) quiere decir ‘polimorfismos nucleotídicos simples’. Es la diferencia de un solo aminoácido entre cerdos individuales en el código genético, o secuencia de ADN. Los SNP son las variaciones genéticas más comunes.
