La revolución genómica ha sido posible gracias a las tecnologías de secuenciación conocidas como de ‘nueva generación’ o de ‘alto rendimiento’, que se esbozaron en el reciente artículo ‘¿A qué nos referimos cuando hablamos de revolución genómica?’ Los principios de la selección genómica son:
- El ADN se analiza mediante un chip para identificar diferencias en el código genético entre animales llamadas polimorfismos de nucleótido único (“Single Nucleotide Polymorphisms”, SNP).
- Los SNP's relacionan segmentos específicos del ADN con diferencias productivas para determinados carácteres en una población concreta.


Uno de los potenciales a largo plazo de la selección genómica es la identificación de genes epecíficos o regiones genómicas con un impacto significativo en la salud porcina y que permita mejorar la selección para resistencia y/o tolerancia a enfermedades. Algunos grandes proyectos y colaboraciones de investigación están trabajando en estas áreas y sus resultados van agrandando la base de datos existente en la literatura científica.
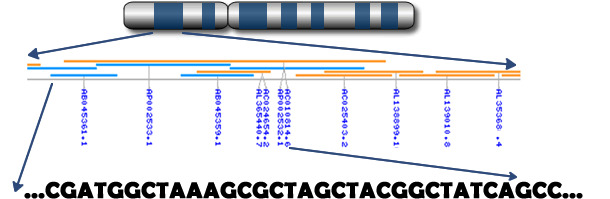
Figura 2. La genómica está identificando SNPs para carácteres de salud (Fuente: Prof. C. Haley – Roslin Institute, Edinburgh)
Por ejemplo, en la reciente edición de la influente PAG (International Plant and Animal Genomics) a principios de este año, entre los principales artículos presentados estaban los siguientes:
- Investigadores de EEUU han identificado tres marcadores que están significativamente asociados con lesiones pumonares causadas por pleuroneumonía y/o neumonía enzootica. Se espera que esto permitirá identificar la variación genética relacionada con el aumento de la resistencia a las enfermedades respiratorias.
- Científicos chinos han trabajado sobre los mecanismos que explican la diferencia en el estatus sanitario entre cerdos Landrace (susceptibles) y Tongcheng (una raza china, que es resistente a la exposición a PRRSV). Han observado la expresión diferenciada del gen CD169 en los cerdos Tongcheng en comparación con los Landrace tras la infección por PRRSV. Algunos estudios presentados muestran que el receptor CD169 mejora la presentación del antígeno a las célulasT.
- El PRRS Host Genetics Consortium (PHGC) fue creado por grupos de investigación de Canadá y EEUU para determinar el papel de la genética en la resistencia a la infección por PRRSV y los efectos relacionados con el crecimiento. Los últimos resultados de estudios en lechones en transición han identificado una región genómica en el cromosoma 4 (SSC4) con un impacto significativo sobre la variabilidad de la carga viral y el crecimiento tras la exposición al virus. Análisis posteriores y la secuenciación de la región SSC4 deberían identificar marcadores que permitan diferenciar entre animales resistentes a PRRSV/con crecimiento máximo de los susceptibles a PRRSV/con crecimiento reducido.
- Otros proyectos del PHGC están estudiando el transcriptoma de los cerdos expuestos a PRRSV. Para que los genes produzcan proteinas, el ADN se transcribe a un RNA mensajero (mRNA). El término transcriptoma incluye todo el mRNA que se utiliza para la producción de proteínas. Este estudio proporcionará más datos para descifrar los mecanismos genéticos de la respuesta del hospedador frente a la infección por PRRSV.
- Investigadores de Iowa State presentaron un análisis genómico que reveló diferencias de expresión en muchos genes entre cerdos clasificados como excretores de salmonela persistentes o moderados. Concluyeron que las diferencias cuantitativas en los valores de IFN-γ explican la expresión de la mayoría de los genes analizados, y que el regulón IFN-γ es una fuente de genes cuyo nivel de expresión dos días post-infección puede predecir la capacidad excretora del animal. Estos genes pueden ser evaluados como candidatos para el desarrollo de análisis predictivos sobre la capacidad excretora de los animales.
- Investigadores chinos han estudiado la respuesta inmune en lechones para 18 caracteres hematológicos, 7 linfocitarios y 3 de las citoquinas utilizando una vacuna contra peste porcina en dos genotipos distintos. Identificaron 12 SNPs significativos correlacionados con la respuesta inmunitaria, lo que sugiere una base preliminar para una posterior identificación de las mutaciones causales subyacentes a la respuesta inmune general.
- Investigadores de Nebraska trabajaron con razas cruzadas infectadas experimentalmente con PCV2b para identificar las principales variaciones genéticas que influyen sobre los principales indicadores de la progresión de la enfermedad y la respuesta inmune frente a PCVAD. Durante un periodo de 28 días de exposición al virus, se midió semanalmente la GMD, la carga viral y los valores de anticuerpos específicos. Los resultados iniciales mostraron variación entre hospedadores en cuanto a magnitud y tiempo de respuesta inmune frente a PCV2b. Los principales clústers de SNPs que afectaron la carga viral estaban localizados en múltiples cromosomas incluyendo SSC6, SSC7 y SSC12. La región localizada en SSC7 también afectó a la ganancia de peso durante la exposición. El clúster de genes para el antígeno leucocitario porcino clase II se localiza en dicha región y puede explicar gran parte de la variación fenotípica observada.

Figura 3. PCVAD – un objetivo clave futuro para la selección genómica (Foto: J. Mackinnon).
Además de los estudios citados, se están investigando los genes/SNPs involucrados en otras infermedades relevantes como la fiebre aftosa, la PPA y la gripe porcina. Sin embargo, la selección futura para una mejora en la salud requierirá el uso de múltiples marcadores y la disponibilidad de grandes bases de datos de ADN de varias poblaciones bien caracterizadas tanto genética como fenotípicamente en las que puedan identificarse asociaciones entre SNPs y caracteres de salud. El próximo artículo de esta serie, ‘¿Cúando dará fruto la genómica?’, analizará esto con más detalle.



