Búsqueda de "Quantitative Trait Loci" (QTL) en porcino: El QTL del cromosoma 4 para engrasamiento y composición de ácidos grasos
15-jul-2003 (hace 21 años 9 meses 4 días)
La utilización de técnicas relacionadas con la biología molecular en el campo de la genética porcina ha permitido comprender con mayor profundidad la arquitectura genética de numerosos caracteres de interés productivo. En este sentido, se ha verificado que una parte muy significativa de la varianza fenotípica para dichos caracteres viene explicada por la existencia de un número reducido de genes o loci con un efecto cuantitativo detectable (quantitative trait loci o QTL). El diseño experimental de los proyectos de búsqueda de QTL en porcino implica la generación de un material animal a partir del cruce de dos razas muy divergentes para los caracteres estudiados. A partir del cruce de las dos razas parentales (F0), se produce una generación híbrida F1 que es entrecruzada para dar lugar a una segunda generación F2. A continuación, se obtienen registros fenotípicos de todos los individuos de la F2 para los caracteres estudiados. Simultáneamente, los individuos F2 son genotipados para una batería de marcadores genéticos microsatélites (aproximadamente un centenar) escogidos de acuerdo a su polimorfismo y su posición en el genoma. Métodos estadísticos como la regresión de Haley-Knott (1992) permiten detectar si existen asociaciones significativas entre el polimorfismo de alguno de los microsatélites analizados y la variación cuantitativa de los diversos registros fenotípicos obtenidos. En el caso de que exista una asociación significativa, se considera que en la vecindad del microsatélite existe un QTL (ver Figura 1). El paso siguiente consiste en identificar el gen/genes que afectan de forma significativa al carácter estudiado. Este proceso es muy costoso, ya que los QTL suelen abarcar regiones genómicas de gran tamaño (10-20 Megabases). Por ello, aunque se han publicado numerosos trabajos sobre QTL, en muy pocos casos se ha llegado a identificar el gen responsable. Es posible que el proyecto de secuenciación del genoma del cerdo contribuya a resolver este problema mediante la identificación precisa del catálogo de genes que caracterizan a dicha especie.
Se han descrito numerosos QTL en porcino para diversos caracteres relacionados con el crecimiento, el engrasamiento, la calidad de la canal, la calidad de la carne, la prolificidad, el número de tetinas etc. Sin embargo, no en todos los trabajos relacionados con un mismo grupo de carácteres se describen los mismos QTL. Esta falta de consistencia puede atribuirse al hecho de que un QTL sea específico de una población o raza determinada, o bien a la existencia de factores relacionados con el diseño de cada experimento en particular. Uno de los QTL detectados de forma más consistente en diversos cruces porcinos es el QTL del cromosoma 4 para engrasamiento y composición de la grasa. Dicho QTL fue descrito por primera vez por Andersson et al. (1994) mediante un cruce de jabalí x Large White. Estaba situado en el intervalo 0-68 centiMorgan (cM) y explicaba un 18% de la variación fenotípica para grasa dorsal y abdominal. El alelo del QTL procedente del jabalí estaba asociado a una menor tasa de crecimiento y a un mayor engrasamiento, tal como era de esperar al no ser una especie seleccionada para dichos caracteres. Posteriormente Knott et al. (1998) y Marklund et al. (1999) describieron la existencia de QTL para engrasamiento utilizando el mismo tipo de cruce y en un intervalo similar. La existencia de este QTL, denominado FAT1, fue confirmada en: (a) otros tipos de cruces entre razas divergentes p.e. Large White x Meishan (Walling et al. 2000, Bidanel et al. 2001), Ibérico x Landrace (Pérez Enciso et al. 2000) y Berkshire x Yorkshire (Malek et al. 2001), y (b) en poblaciones comerciales no cruzadas de las razas Large White, Hampshire y Pietrain (Evans et al. 2003, en prensa).
Asimismo, se ha verificado que el QTL FAT1 explica el 25-40% de la variación fenotípica del porcentaje de ácido linoleico en tejido subcutáneo adiposo (Pérez Enciso et al. 2000). Igualmente, Clop et al. (2003) hallaron QTL para para el porcentaje de ácido linoleico, el índice de peroxidabilidad y el índice de dobles enlaces en el cromosoma 4, aunque su significación disminuía de forma notable al utilizar el espesor del tocino dorsal como covariable en el modelo de regresión.
La futura identificación del gen del QTL del cromosoma 4, cuyas variantes alélicas están asociadas a un mayor o menor engrasamiento, y la optimización de un método de genotipado resultaría de gran utilidad para la industria porcina, ya que permitiría predecir de forma muy fiable el valor genético de un individuo para este carácter. Asimismo, y desde una perspectiva a más largo plazo, la combinación de distintas aproximaciones basadas en la identificación de QTL, la secuenciación completa del genoma porcino y la realización de estudios funcionales y de expresión génica, permitirá elaborar un catálogo de genes cuyo polimorfismo afecte a distintos caracteres de importancia productiva. Este conocimiento resultaría fundamental para poder valorar directamente, sin que se produzcan interferencias de tipo ambiental, el genotipo de un individuo para todos aquellos caracteres que sean objeto de selección en las explotaciones porcinas.
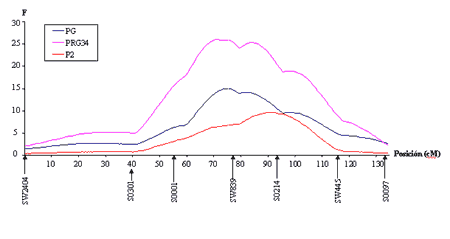 |
Figura 1. Imagen del QTL del cromosoma 4 para los caracteres peso de la grasa dorsal (PG), espesor de la grasa dorsal medido con sonda óptica Fat-O-meter (P2) y espesor de la grasa dorsal medido a las 24 h post-mortem con regleta (PRG34) (según Clop 2001). En el eje de abscisas se indica la posición, expresada en centiMorgan (cM), de cada uno de los marcadores genéticos microsatélites analizados, mientras que en el eje de ordenadas se indica la significación estadística F de cada QTL. Puede observarse que la máxima significación del QTL se alcanza en el intervalo definido por los marcadores S0001 y S0214. Los resultados obtenidos permiten predecir la existencia de un gen o genes, localizados entre ambos marcadores, cuyo polimorfismo tiene un efecto directo sobre la variación cuantitativa de PG, P2 y PRG34.
