Detección laboratorial del virus de PRRS
26-feb-2015 (hace 10 años 1 meses 7 días)
Detección de antígenos virales de PRRS
Un diagnóstico definitivo de PRRS en cerdos enfermos se puede lograr mediante la detección de lesiones microscópicas características de PRRSv junto con la demostración de antígeno viral en los tejidos con lesiones. Para detectar antígeno(s) de PRRSv en tejidos se puede utilizar fluorescencia de anticuerpos (FA) o inmunohistoquímica (IHC) en secciones de tejido congelado (figura 1). Estas pruebas se combinan con anticuerpos monoclonales o policlonales específicos para PRRSv. La prueba de FA directa en secciones de tejido congelado es barata y rápida. Es específica pero no siempre es muy sensible. Los resultados se ven especialmente afectados por la calidad del tejido (p. ej., autolisis). En cambio, la IHC es útil para detectar antígeno viral en tejidos fijados con formalina. La IHC es más sensible que la FA directa, pero conlleva más tiempo y es más cara que la FA.
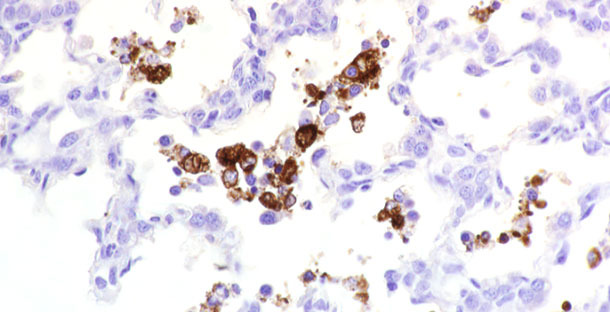
Figura 1. Antígeno de PRRSv detectado en tejido.
Para la FA directa deben enviarse tejidos frescos o congelados y, si se envían para IHC, tienen que fijarse en formalina neutra tamponada al 10%. Sin embargo, la fijación prolongada de tejidos en formalina es perjudicial para una detección eficaz de antígenos mediante IHC. Si se espera un retraso en la realización de la prueba, se recomienda colocar los tejidos en alcohol tras la fijación con formalina. Los tejidos preferidos para estas pruebas son corazón, riñón, pulmón, linfonodos, bazo, timo y amígdalas. Los antígenos de PRRSv también se pueden detectar en la glándula adrenal, intestino, hígado y, en ocasiones, en cerebro. Al realizar FA y IHC, se pueden utilizar anticuerpos monoclonales específicos para antígenos altamente conservados entre los aislados de EEUU y Europa, o se pueden usar anticuerpos monoclonales contra epítopos menos conservados para evaluar las diferencias antigénicas entre cepas. Si el diagnóstico de PRRS se realiza con FA o IHC, debido a la alta especificidad de los anticuerpos monoclonales y la variabilidad antigénica significativa del virus de PRRS, puede ser mejor usar más de un anticuerpo monoclonal en la prueba para evitar un diagnóstico erróneo.
Detección de material genómico viral de PRRS
Se han desarrollado pruebas basadas en la reacción en cadena de la polimerasa (PCR) para detectar el ARN de PRRSv (es decir, ácido nucleico) en muestras clínicas. Dado que el virus no necesita ser aislado en cultivo celular para detectar el ARN viral, PCR puede proporcionar de forma más rápida el resultado de la prueba que el aislamiento del virus. Como principio general, se considera que los ensayos basados en PCR son muy sensibles y muy específicos.
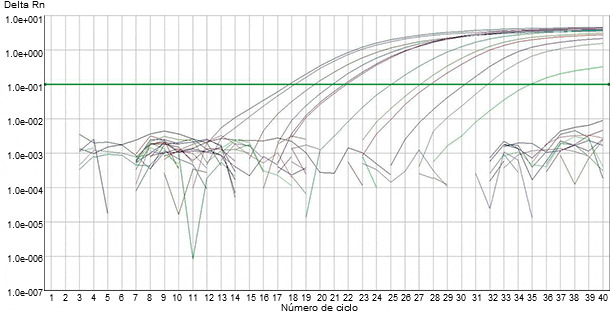
Figura 2. Detección de material genómico viral de PRRS en muestras clínicas mediante PCR fluorogénica automatizada.
Se han desarrollado varios tipos de análisis basados en PCR que utilizan cebadores específicos, complementarios a las secuencias de diferentes fragmentos genómicos, para la detección de materiales genéticos virales de PRRS en muestras clínicas. En cualquier análisis basado en PCR para PRRSv, el ARN viral necesita ser extraído de las muestras clínicas y transformado en ADN complementario (ADNc) usando una enzima transcriptasa inversa (RT). Posteriormente, el ADNc se amplifica exponencialmente mediante la PCR usando una enzima Taq polimerasa y cebadores específicos del virus. La coincidencia exacta de los cebadores, seguida por la amplificación exponencial, está destinada a producir un test muy sensible y muy específico. Recientemente se han desarrollado tests basados en la PCR fluorogénica automatizada (es decir, RT-qPCR) para detectar material genómico viral de PRRS en muestras clínicas (figura 2). Estas PCR emplean la amplificación en un solo paso en un tubo en el que sondas fluorescentes se unen al producto de la PCR a medida que se produce (es decir, PCR "en tiempo real"). Se cree que las PCR fluorogénicas mejoran la fiabilidad y la consistencia de las RT-PCR convencionales para detectar PRRSv (figura 3).
Figura 3. PCR fluorogénica para detectar PRRSv.
Las pruebas basadas en PCR se han utilizado para detectar ARN de PRRSv en diferentes muestras clínicas, como fluido oral y muestras ambientales. La PCR es particularmente útil para la detección de ARN viral en muestras de semen o heces, que son citotóxicas para cultivo celular o no se pueden evaluar por otros métodos. También es útil para detectar ARN de PRRSv en tejidos fetales y fluidos torácicos, es decir, muestras donde PRRSv puede inactivarse fácilmente por autolisis. El uso de análisi de PCR se ha vuelto más común tanto para el diagnóstico de PRRS como para ayudar en la monitorización/vigilancia (es decir, en la selección de animales de reposición, detección de animales portadores y programas de prueba y eliminación) o en la bioseguridad de la explotación (es decir, pruebas ambientales, comprobación de camiones, pruebas de semen).
La PCR es una prueba cara en comparación con otros métodos de diagnóstico. Además, debe tenerse en cuenta que un resultado positivo de PCR indica la presencia de ARN viral pero no necesariamente la presencia de PRRSv infeccioso. Además, la realización de pruebas de PCR en diferentes laboratorios puede variar dependiendo de la condición de la muestra, procesamiento de la muestra, técnica de extracción, cebadores utilizados, condiciones de ciclos térmicos y la habilidad y experiencia del técnico que realiza el ensayo. Por lo tanto, es importante que los laboratorios que realizan la PCR para validar sus ensayos incluyan estimaciones de sensibilidad, especificidad, comparaciones con los estándares de referencia, resultados de ensayos de aptitud y estudios experimentales o de campo del ensayo.
