Diagnóstico de infección por Escherichia coli en cerdos: tipificando aislados de E. coli
12-feb-2015 (hace 10 años 1 meses 22 días)
E. coli (EC) – cultivo y serotipificación
E. coli es una bacteria gram-negativa en forma de bacilo que tiene multitud de variantes. Además de cepas comensales, que de manera natural se encuentran en la flora intestinal de todos los mamíferos, también existen cepas patógenas clasificadas como E. coli intestinal (InPEC) o extraintestinal (ExPEC). Sin embargo, la diferenciación de estas variantes ha planteado durante mucho tiempo un problema de diagnóstico debido a que diferentes patotipos o patovares no pueden ser identificados con métodos fenotípicos sencillos.
E. coli puede detectarse en cerdos con relativa facilidad mediante el cultivo en agar sangre (figura 1) o medios diferenciales (p. ej., agar Gassner o MacConkey) de muestras adecuadas (intestino delgado, heces) de animales con infección aguda. Años atrás se había utilizado con éxito la tipificación de serotipos o serovares para diferenciar aislados de E. coli, debido a que ciertos serotipos se asocian frecuentemente con determinadas enfermedades (tabla 1). Por ello, en veterinaria es habitual determinar los antígenos O y F, por aglutinación rápida en placa utilizando sueros poli- o monovalentes (los antígenos O son componentes de los LPS de la pared celular bacteriana y son también endotóxicos; los antígenos F [fimbrial] son adhesinas que facilitan la adhesión huésped-específica para colonizar las células epiteliales del intestino delgado). Aunque este método es rápido, está sujeto a errores por reacciones cruzadas, enmascaramiento de antígenos diana o incapacidad de las fimbrias para desarrollarse in vitro, lo que puede conducir a clasificaciones erróneas. Por otro lado, desde hace décadas la serofórmula O:K:H ha sido y sigue siendo el estándar para una determinación completa de todos los serotipos, p. ej., para determinar no sólo los antígenos O, sino también los K (polisacárido capsular) y H (proteína flagelar).
Tabla 1: Patógenos E. coli intestinales en cerdos
| Patotipos |
Enfermedades causadas por E. coli |
Serotipos de antígeno O frecuentemente asociados | Serotipos fimbriales frecuentemente asociados |
| E. coli patógeno intestinal (InPEC) | Diarrea por E. coli en lechones | O8, O108, O138, O139, O141, O147, O149 y O157 | F4, F5, F6 y F41 |
| Diarrea por E. coli en cerdos destetados | O8, O108, O138, O139, O141, O147, O149 y O157 | F4, F18ac | |
| Enfermedad de los edemas en cerdos destetados | O138, O139 y O141 (O147, O157 y rutinariamente no serotipables) | F18ab |
E. coli (EC) – patotipos y virotipificación
Otros avances en métodos de biología molecular y mejoras en el análisis biológico molecular de E. coli han llevado a la identificación de muchos genes que codifican factores de virulencia (tabla 2): por un lado, factores de virulencia clásicos con efectos nocivos (toxinas) y, por el otro, aquellos que directa o indirectamente facilitan la colonización y la supervivencia de E. coli en el hospedador. La identificación de estos genes, por ejemplo, por PCR o técnica array, hace que ahora sea posible determinar la virulencia potencial de aislados de E. coli y distinguir entre diferentes patotipos (tabla 3). La identificación de estos marcadores de virulencia y, por lo tanto, de sus genes diana se llama virotipificación.
Tabla 2: Ejemplos de genes diana detectados en E. coli y los factores de virulencia
(fimbrias, adhesinas, toxinas) para los que codifican
| Genes diana | Factores de virulencia |
| faeG (F4) | Fimbrias F4 |
| fanC (F5) | Fimbrias F5 |
| fasA (F6) | Fimbrias F6 |
| fedA (F18) | Fimbrias F18 |
| fim41A (F41) | Fimbrias F18 |
| fimH (F1) | Fimbrias tipo 1 |
| fimA (F1) | Fimbrias tipo 1 |
| papC | Fimbrias P |
| aidA (AIDA) | Adhesina autotransportadora AIDA-I |
| paa | Factor de adherencia porcino |
| eaeA (intimina) | Intimina |
| eltB (LTI) | Enterotoxina sensible al calor |
| estA (STI) | Enterotoxina resistente al calor |
| est B (STII) | Enterotoxina resistente al calor |
| estA (EAST) | Enterotoxina resistente al calor |
| stx2e | Shigatoxina variante 2e |
| cdtB | Toxina citoletal distensora |
| cnf1 | factor citotóxico necrotizante tipo 1 |
| iucD | Aerobactina |
| escV | Sistema de secreción tipo III |
| pic | Autotransportador serin proteasa |
Tabla 3: Genes diana para la identificación de patotipos de E. coli en cerdos
| Patotipos | Fimbrias y adhesinas (genes diana) | Toxinas (genes diana) | Síntomas | |
| E. coli patógeno intestinal (InPEC) |
E. coli enteropatogénica (EPEC)
|
fimA/fimH paa intimina |
escV pic cdtB |
diarrea por malabsorción |
|
E. coli enterotóxica (ETEC) |
F4, F5, F6, F18, F41 fimA/fimH AIDA paa |
STI, STII, EAST LTI Stx2e hemolisina |
diarrea por hipersecreción | |
| Enfermedad de los edemas por E. coli (EDEC) | F18 AIDA |
Stx2e hemolisina |
enfermedad de los edemas | |
| E. coli formadora de Shigatoxina (STEC) | fimA, fimH intimina |
Stx2e EAST escV pic, cdtB hemolisina |
diarrea catarral-hemorrágica | |
| E. coli patógeno extraintestinal(ExPEC) |
E. coli septicémica (SEPEC) |
papC | iucD | septicemia hemorrágica |
| E. coli uropatogénica (UPEC) | papC fimA, fimH | cnf1, iucD hemolisina |
mastitis por coliformes en cerdas | |
No obstante, los aislados de E. coli patógeno siempre contienen una combinación de fimbrias y adhesinas, además de toxinas. Varias toxinas y adhesinas pueden estar presentes a la vez, y en diferentes combinaciones. La determinación de estos diversos marcadores de virulencia y la identificación de sus genes proporciona información sobre el patotipo de E. coli aislado del cultivo celular (tabla 3). Hay que destacar que los E. coli hemolizantes no son los únicos que son patógenos (figura 1), y de hecho los E. coli no hemolizantes, en particular, son determinados con frecuencia como causa de enfermedad clínica. Por otra parte, los E. coli hemolizantes no suelen poseer genes relevantes para fimbrias y toxinas. Por lo tanto, si es posible, todas las variantes de aislados de E. coli identificadas por cultivo deben virotipificarse con el fin de asegurar que el patógeno responsable puede también ser identificado. La información sobre los tipos y toxinas fimbriales de E. coli patógeno determinados también es útil en la selección de vacunas adecuadas con los antígenos correspondientes.
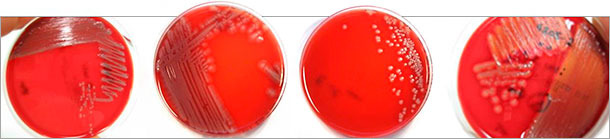
Figura 1: Diferentes morfologías coloniales de aislados de E. coli patógenos en agar sangre (Columbia) identificados tras virotipificación por PCR múltiple. No sólo los E. coli hemolizantes (derecha) son patógenos.
En la interpretación de los resultados de diagnóstico hay que tener en cuenta que los patotipos de E. Coli también pueden aislarse en huéspedes sanos (infección latente), sobre todo de la flora intestinal. Por lo tanto, la evaluación de los resultados sólo puede hacerse teniendo en cuenta tanto los hallazgos clínicos en el animal como la concentración de los patógenos aislados con el patotipo identificado.
La interpretación del virotipificado se vuelve complicada cuando los marcadores de virulencia se detectan directamente en el material de la muestra, p. ej. heces, sin necesidad de tipificar los aislados individuales. Aunque esto es técnicamente posible y se realiza bajo demanda, cuando diferentes patotipos de E. coli están presentes en la muestra, también se obtiene una mezcla de todos los factores detectables, que puede conducir a la identificación de falsos patotipos y una combinación de marcadores de virulencia que no existe como tal. Además, también es posible que se detecten marcadores de virulencia de otras bacterias intestinales presentes en la muestra de heces, ya que estas bacterias tienen genes similares. En este caso, se detectarán posibles marcadores de virulencia de E. coli, que de hecho no son factores de virulencia para el E. coli detectado en la muestra. Por lo tanto, se debe tener un especial cuidado en la interpretación de los resultados obtenidos en el virotipificado de muestras.
En combinación con el examen clínico y el a menudo necesario examen patológico (descrito con anterioridad) y con diagnósticos diferenciales apropiados, el virotipificado de los aislados de E. coli proporciona una notable mejora en el diagnóstico de infecciones por E. coli en el cerdo.
