Microbiota y producción porcina: Cómo interpretar los estudios de la microbiota - Parte 2
12-sep-2022 (hace 2 años 6 meses 19 días)
En el artículo anterior se destacó la dificultad de realizar estudios científicamente válidos sobre la microbiota porcina. Sin embargo, hemos visto un número cada vez mayor de estudios publicados sobre este tema (figura 1). A medida que disponemos de más información, ¿Cómo podemos dar sentido a todos estos datos y aplicarlos en la granja?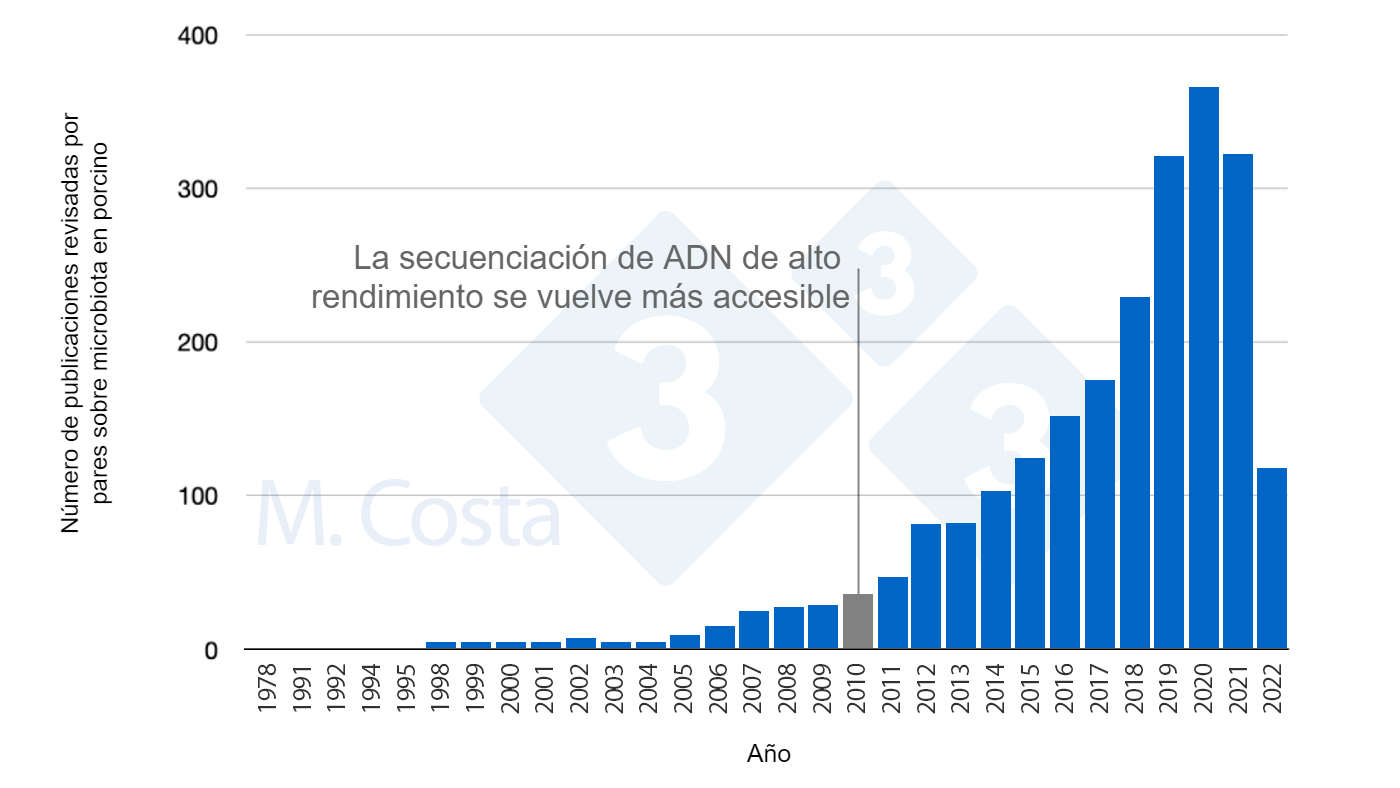
Paso 1 – Conoce tus condiciones
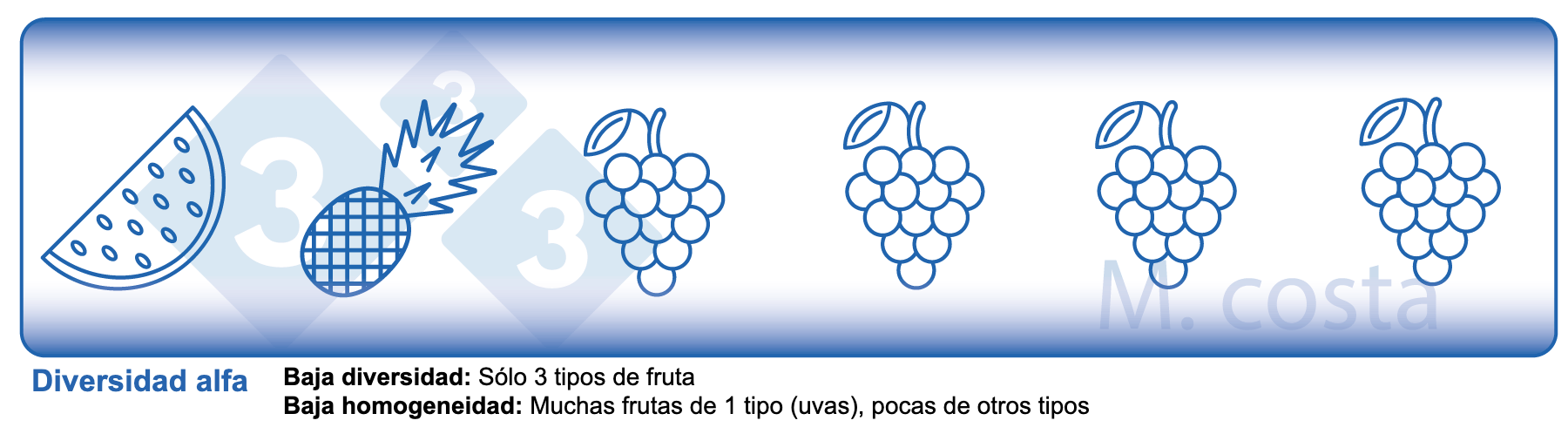
Estos son algunos de los parámetros clave usados en los estudios sobre microbioma que facilitan la comprensión de los resultados. Muchos de ellos adquiridos de la ‘’macro’’ ecología, después de todo, la comunidad bacteriana en el intestino no es muy diferente de un banco de peces en el mar. Ambos tienen relación con su entorno y con otros seres con los que comparten su espacio/hábitat. Por lo tanto, entender estos parámetros permite interpretar correctamente los experimentos centrados en el microbioma. En general, este tipo de estudios se centran en aclarar qué microbios están presentes/ausentes en una muestra: responden a la pregunta general de ¿Quién está ahí? y ¿Cuántos hay?. Las diferentes especies/tipos de microbios en un hábitat determinado (por ejemplo, el intestino) suelen referirse a la diversidad de la comunidad. Esta diversidad de organismos (por ejemplo, frutas o bacterias) en un único hábitat (por ejemplo, las heces del lechón nº 495 el tercer día post destete) se denomina diversidad alfa (figura 2) y tiene en cuenta dos aspectos principales:
- Diversidad de especies: cuántos tipos de diferentes bacterias/especies hay.
- Homogeneidad de especies: la distribución (abundancia) de estas especies, o si un organismo es predominante (más abundante) que otros.
Existen muchos índices utilizados para medir la diversidad alfa, los más comunes son el índice de Shannon, el de Chao1 o el de Simpsom. Cada uno de estos mide diferentes aspectos de la diversidad alfa. Estos índices se utilizan frecuentemente en los estudios del microbioma debido a que reflejan la diversidad en la muestra.



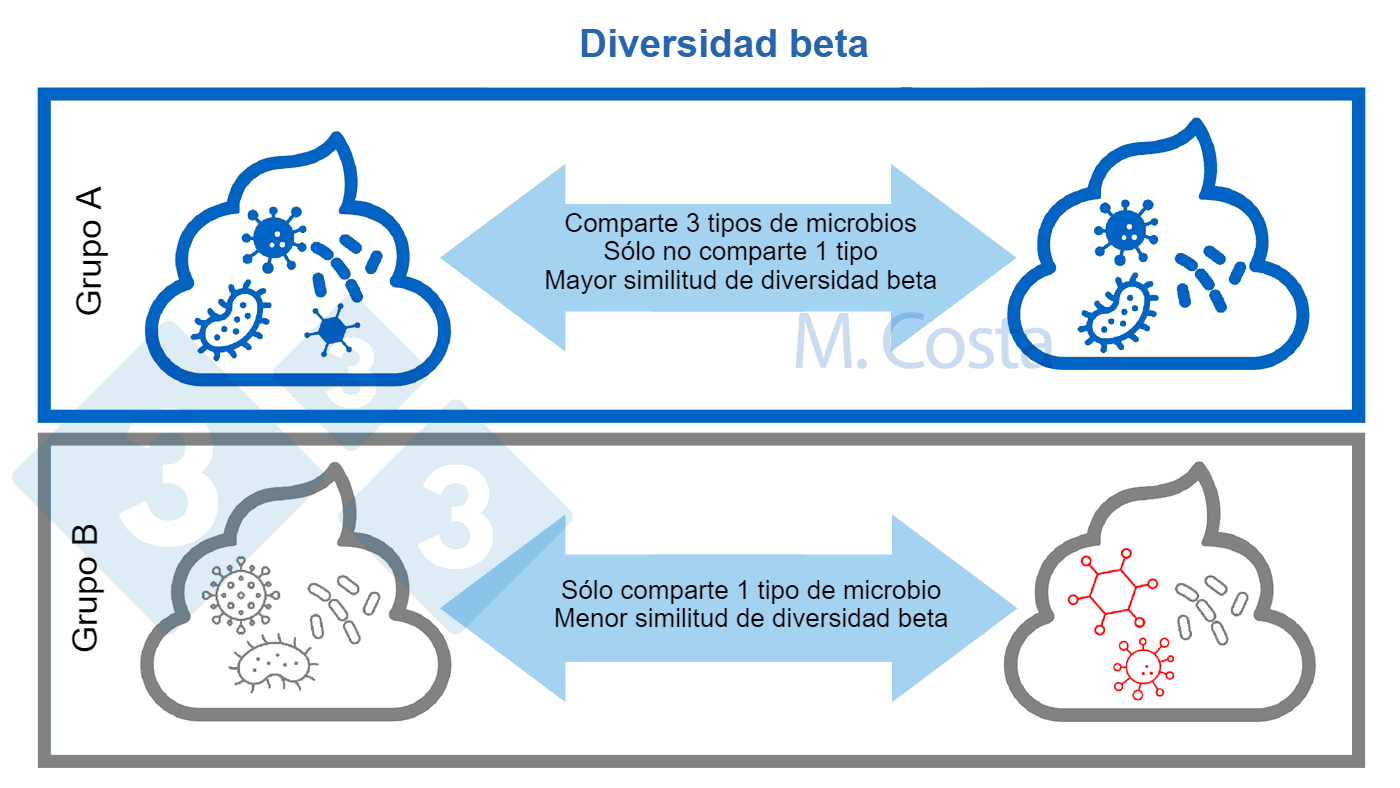
Cuando comparamos la comunidad bacteriana de dos hábitats (por ejemplo, muestras fecales de cerdos tratados con antibióticos o no tratados), investigamos su diversidad beta o la similitud entre muestras. ¿Tienen estas comunidades muchas cosas en común (como por ejemplo, podemos encontrar las mismas bacterias en la muestra A y B, figura 3)? ¿Son similares o distintas? De nuevo, existen muchos índices para medir esto, como el Unifrac, el Jaccard, la disimilitud de Bray-curtis etc. Éstos tienen en cuenta diferentes aspectos de la diversidad de la comunidad, como pueden ser la relación (filo)genética entre microbios o la abundancia de cada muestra.
En general, la diversidad alfa de la microbiota intestinal tiende a aumentar y la diversidad beta a disminuir a lo largo de la vida del cerdo (figura 4).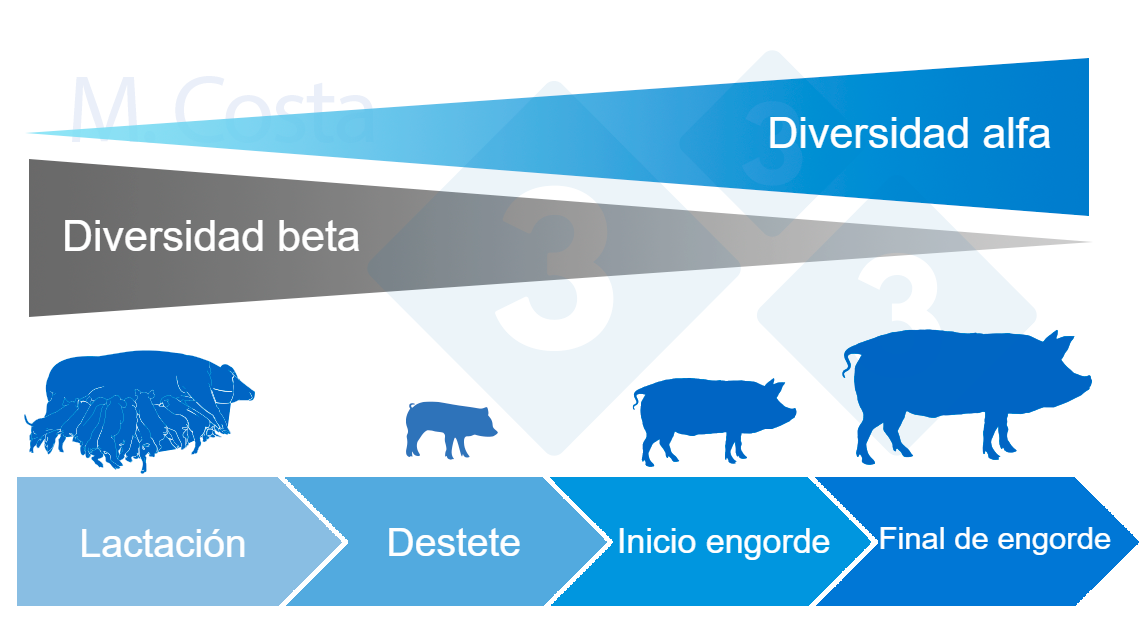
Paso 2 - ¿El estudio incluye los controles adecuados?
Cuando se trata de estudios del microbioma, todos los esfuerzos se deben centrar en minimizar el efecto de los contaminantes. El ADN microbiano está literalmente en todas partes (incluso si los microbios están muertos, su ADN sigue ahí) y las tecnologías de secuenciación del DNA actuales pueden detectar pequeñas cantidades (como se comentó en la parte 1). Así pues, los estudios no sólo deben incluir los grupos experimentales apropiados (por ejemplo, cerdos tratados y no tratados con antibióticos del mismo lote, alimentándose con la misma dieta...), también los grupos control adecuados (tubos de recogida vacíos o kits de secuenciación sin muestra) para ayudar a identificar los contaminantes y eliminarlos del análisis. Si no se muestran en el estudio, cualquier conclusión podría ser errónea.
Paso 3 - Importancia biológica y más allá del valor P (o: ¿Esto realmente afecta a los animales?)
Es fácil alterar la estructura de la comunidad microbiana. La microbiota intestinal, por ejemplo, es susceptible a los cambios en la dieta, el espacio físico y los antibióticos. Las intervenciones pueden provocar fácilmente cambios significativos en la composición de la comunidad (diversidad alfa y beta). En las últimas décadas, se han realizado importantes trabajos para confirmarlo. Sin embargo, la evidencia de la importancia biológica es la clave. Mucho del ''trabajo'' de los microbios es redundante. Por ejemplo, muchos organismos de la microbiota intestinal pueden producir los mismos ácidos grasos de cadena corta. Por esto, cambios en la comunidad microbiana (o ''¿Quién está ahí?) es probablemente menos importante que lo que están haciendo. Es importante buscar pruebas indirectas de que esto último se haya afectado por la intervención, y puede hacerse de diferentes maneras: tasa de crecimiento, resistencia a enfermedades o función de la barrera intestinal. Realmente depende del estudio, pero debe ser descrito.
Como ocurre con cualquier otra tecnología de vanguardia, algunos aspectos importantes se pueden perder al trasladar la investigación del laboratorio a la granja. A medida que la modulación del microbioma y sus aplicaciones en producción porcina sean más claros, deberíamos ver nuevas estrategias para ayudar a impulsar el rendimiento y la resiliencia a enfermedades.
