Una larga carrera profesional en la investigación de PRRSV
El Dr. Hans Nauwynck, profesor de la Universidad de Gante (Bélgica) tiene una carrera investigadora impresionante y productiva. Es autor de más de 450 artículos revisados por pares, 3 capítulos de libros y 76 tesis en los últimos 30 años. Es considerado uno de los investigadores europeos más importantes, ¡sin duda el más enérgico! En este artículo resumimos nuestras conversaciones sobre cómo el PRRSV ha ido evolucionando en el campo con el tiempo.

Problemas relacionados con PRRSV Tipo 1
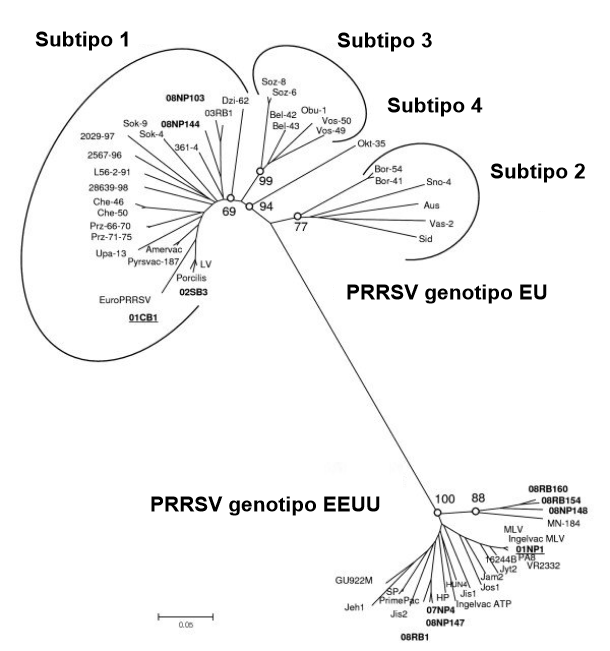
Al principio de su carrera (90’s), realizó experimentos para examinar el comportamiento de las cepas PRRSV-1 y PRRSV-2. Aunque ambos inducían fallos reproductivos en cerdas, eran completamente diferentes a nivel del tracto respiratorio en cerdos jóvenes. La dificultad respiratoria fue evidente con el tipo 2. Las cepas de tipo 1 causaron principalmente infecciones subclínicas. En esos años aprendió que en la UE estábamos lidiando con un virus totalmente diferente del de EEUU/Canadá. La gran distancia genética concordó con los hallazgos clínicos (imagen 1). Las experiencias en el campo también fueron muy diferentes. Con las cepas PRRSV-1, el virus se estaba propagando de forma extremadamente lenta en los engordes. El virus tardaba todo el ciclo del engorde en infectar a todos los cerdos. Esto apuntaba a una potencia de transmisión muy ineficiente. Con PRRSV-2 se experimentó una propagación más rápida. En EEUU, los científicos demostraron una propagación aerógena. En los años 2000’s, se identificaron cepas del tipo PRRSV-1 genéticamente alejadas en Rusia/Bielorrusia (subtipos 2 y 3) que eran mucho más virulentas y patógenas. Más recientemente, el PRRSV-1 subtipo 1 se está extendiendo más rápido e incluso está causando más enfermedad de lo habitual (cepas italianas).
“Europa occidental, no tiene que preocuparse por el PRRSV tipo 1 como patógeno responsable de problemas respiratorios; deben temer y prestar atención a las enfermedades o coinfecciones asociadas”, dice el Dr. Nauwynck. Creó un nombre para los problemas respiratorios relacionados con PRRS en la UE, "PRRS PLUS" (lanzó este término para los veterinarios porcinos de campo). Las múltiples coinfecciones por PRRSV con otros patógenos diferentes hacen del PRRSV uno de los patógenos más importantes en la UE. Como resultado, es esencial tener un análisis diagnóstico completo. Todos los patógenos deben ser identificados. En este contexto, son necesarios nuevos diagnósticos. Es esencial un conocimiento completo de los patógenos circulantes para comprender lo que está sucediendo en la granja y para controlar el “PRRS PLUS”. Como él dice: "Si los veterinarios se encargan de todas las posibles coinfecciones, los signos respiratorios clínicos del PRRS básicamente desaparecen". Él cree que las granjas positivas europeas pueden criar cerdos de manera muy eficiente cuando controlan los cofactores y no necesitan erradicar el virus. Esto puede cambiar, por supuesto, cuando la virulencia/patogenicidad del virus aumente en el futuro.
La evolución del virus puede explicarse por un mejor conocimiento de las interacciones virus-hospedador: el papel de los receptores Siglec
Su equipo se ha volcado en el estudio de la interacción virus-hospedador a nivel molecular y, como resultado, han podido comprender cómo ha ido cambiando el virus con el tiempo, específicamente cómo aumentó su poder de transmisión, como se describió anteriormente, y su patogenicidad.
Su equipo demostró que la entrada del virus en los macrófagos implica múltiples pasos. Comienza con la interacción del virus con los receptores Siglec y continua tras la internalización con la interacción con el receptor CD163, lo que conduce al desmontaje del virus. PRRSV puede utilizar diferentes Siglecs y evolucionar en el tiempo.
Siglec-10 puede ser hipotetizado como el “receptor-arcaico”. Siglec 10 está presente en un pequeño grupo de macrófagos en las amígdalas y otros tejidos linfoides y es muy eficiente en el proceso de entrada del virus, lo que demuestra su larga co-evolución con PRRSV. Este Siglec probablemente fue usado por el virus al comienzo de su aparición. En ese momento, PRRSV solo se estaba replicando en esta pequeña población de macrófagos y probablemente no causaba signos clínicos. Debido a que siglec-10 también está presente en los linfocitos B, pudo unirse y entrar en estas células pero sin infectarlas. Esta podría ser la base de la persistencia del virus en su huésped (reservorio). A finales de los años 80, el PRRSV probablemente cambió adicionalmente al receptor Siglec-1 (= sialoadesina, CD169). Las células positivas para Siglec-1 están presentes en los pulmones, la placenta y los tejidos linfoides. La replicación en los pulmones y la placenta resultó en problemas respiratorios y reproductivos. Esa fue la base del nombre PRRSV. En el 2000, el virus en Europa occidental comenzó a replicarse en los macrófagos nasales, lo que condujo a un aumento de la transmisión entre los cerdos. Se desconoce el receptor responsable de esta adaptación; es diferente al Siglec-1 y Siglec-10. La cepa de Lena altamente virulenta (PRRSV-1 subtipo 3) está causando patología de los vasos, que está asociada con la replicación del PRRSV en los macrófagos venosos. El receptor en este subconjunto de macrófagos también es diferente de Siglec-1 y Siglec-10. Se están investigando los receptores en los macrófagos venosos y nasales.
Cree que el virus sigue evolucionando hacia formas que infectan cada vez más subpoblaciones de macrófagos.








